RESUMO
O objetivo do presente trabalho foi avaliar a variabilidade genética de larvas e alevinos de piracanjuba em programa de repovoamento. Foram coletadas 180 larvas de piracanjuba de três dias e 90 alevinos de três meses de idade. Foram avaliados cinco loci microssatélites, os quais produziram 19 alelos. Não houve presença de alelos raros nem perdas de alelos ao longo do período. A heterozigosidade observada foi superior nas larvas em relação aos alevinos. Houve desvio no equilíbrio de Hardy-Weinberg na maioria dos loci em ambos os grupos. O coeficiente de endogamia foi positivo em ambos os grupos, sendo a média dos alevinos superior em relação às larvas. O excesso de heterozigotos foi significativo no modelo Stepwise Mutation Model para os alevinos, indicando a possibilidade de efeito gargalo recente. Conclui-se que, apesar da adequada variabilidade genética encontrada, os valores do coeficiente de endogamia e a possibilidade de efeito gargalo nos alevinos atentam para a necessidade de constante monitoramento genético desses estoques antes da liberação no ambiente.
Palavras-chave:
conservação; efeito gargalo; monitoramento genético; repovoamento
ABSTRACT
The objective of this study was to evaluate the genetic variability of Piracanjuba larvae and fingerlings in restocking program. A total of 180 three-day Piracanjuba larvae and 90 three-month-old fish were sampled. Five microsatellite loci were evaluated, which produced 19 alleles. There were no rare alleles or loss of alleles over the period. The observed heterozygosity was higher in larvae compared to fingerlings. There was a deviation in Hardy-Weinberg equilibrium in most loci in both groups. The inbreeding coefficient was positive in both groups, with the average of the fingerlings superior to the larvae. The excess heterozygotes were significant in the Stepwise Mutation Model for the fingerlings, indicating the possibility of a recent bottleneck effect. Despite the adequate genetic variability found, the values of the inbreeding coefficient and the possibility of bottleneck effect in the fingerlings show the need for constant genetic monitoring of these stocks prior to release into the environment.
Keywords:
conservation; bottleneck effect; genetic monitoring; restocking
INTRODUÇÃO
O repovoamento de peixes consiste na soltura de espécies em um determinado corpo de água, sendo empregado com o intuito de mitigar ações antrópicas que provocaram reduções dos estoques e que não podem ser removidas ou evitadas (Aprahamian et al., 2003APRAHAMIAN, M.; SMITH, K.M.; McGINNITY, P. et al. Restocking of salmonids-opportunities and limitations. Fish. Res., v.62, p.211-227, 2003. ). A construção de barragens, sobrepesca e contaminação das bacias hidrográficas englobam as principais ações antrópicas que alteram as características químicas e físicas dos corpos de água (Agostinho et al., 2007AGOSTINHO, A.A.; GOMES, L.C.; PELICICE,F.M (Eds.). Ecologia e manejo de recursos pesqueiros em reservatórios do Brasil. Maringá: Eduem, 2007. 501p. ), gerando consequências negativas principalmente para as espécies de migradores, as quais dependem do curso natural do rio para sua reprodução (Murgas et al., 2004MURGAS, L.D.S.; MILIORINI, A.B.; FRANCISCATTO, R.T. et al. Viabilidade espermática do sêmen de piracanjuba (Brycon orbignyanus) resfriado a 4°C. Rev. Bras. Zootec., v.33, p.1361-1365, 2004.) e, portanto, perpetuação da espécie. O repovoamento de peixes é a estratégia mais adotada pelas concessionárias hidrelétricas e entidades responsáveis pelo manejo de reservatórios do Brasil e tem por objetivo diminuir o impacto desses fatores sobre o estoque de peixes nativos das respectivas bacias hidrográficas (Agostinho et al., 2007).
Fatores como espécie, sanidade, estrutura genética e adaptabilidade devem ser levados em consideração ao se realizar o repovoamento, a fim de evitar efeitos negativos e irreversíveis sobre a população selvagem de peixes. A manutenção da variabilidade genética é um aspecto fundamental nesse processo, uma vez que a maioria dos programas de repovoamento utiliza peixes de cativeiro, os quais são normalmente sustentados por muitas gerações (González-Wangüemert et al., 2012). Dessa forma, faz-se necessário o monitoramento tanto dos reprodutores utilizados na produção dos estoques quanto dos indivíduos que serão soltos nos rios, para garantir a variabilidade genética deles e evitar efeitos na ictiofauna devido à deriva genética e consequente diminuição da capacidade de adaptação ao novo ambiente (Rodriguez-Rodriguez et al., 2010; Lopera-Barrero et al., 2016; Ribeiro et al., 2016RIBEIRO, R.P.; RODRIGUEZ-RODRIGUEZ, M.P.; RESENDE, E.K. et al. Genetic characteristics of Tambaqui broodstocks in the state of Rondônia, Brazil: implications on production and conservation. Semin. Cienc. Agrar., v.37, p.2375-2386, 2016. ).
A piracanjuba (Brycon orbignyanus) é um peixe nativo das bacias hidrográficas Uruguai e Paraguai e desperta interesse comercial em razão de seu rápido crescimento, carne saborosa e apreciação na pesca esportiva (Borba et al., 2006BORBA, M.R.; FRACALOSSI, D.M.; PEZZATO, L.E. Dietary energy requirement of piracanjuba fingerlings, Brycon orbignyanus, and relative utilization of dietary carbohydrate and lipid. Aquacult. Nutr., v.12, p.183-191, 2006. ; Maria et al., 2006MARIA, A.N.; VIVEIROS, A.T.M.; FREITAS, R.T.F. et al. Extenders and cryoprotectants for cooling and freezing of piracanjuba (Brycon orbignyanus) semen, an endangered Brazilian teleost fish. Aquaculture, v.260, p.298-306, 2006.). Ela se encontra ameaçada de extinção (Machado et al., 2008MACHADO, A.B.M.; DRUMMOND, G.M.; PAGLIA, A.P. (Eds.). Livro vermelho da fauna brasileira ameaçada de extinção. Brasília: MMA, 2008. 1420p. ), devido principalmente à construção de barragens, as quais afetam seu processo de migração reprodutiva, bem como pela destruição da vegetação ripária, redução da alimentação natural, poluição e sobrepesca (Panarari-Antunes et al., 2011). Segundo esses autores, com a diminuição dos estoques dessa espécie, ocorre a perda da variabilidade genética, o que causa maior vulnerabilidade dela.
Devido a pressões seletivas, como ambientação, mortalidade e canibalismo nas fases iniciais de vida, já foram demonstradas diferenças entre variabilidade genética das larvas e alevinos dentro do mesmo lote de um programa de repovoamento (Gomes et al., 2011GOMES, P.C.; RIBEIRO, R.P.; SIROL, R.N. et al. Diversidade genética de dourado utilizado em programas de repovoamento no rio Paranapanema. Pesqui. Agropecu. Bras., v.46, p.167-173, 2011. ). No entanto, estudos que avaliam a variabilidade genética dos alevinos que serão soltos no ambiente e o efeito da idade sobre a variabilidade genética de piracanjuba ainda são escassos. Dessa forma, o objetivo deste trabalho foi avaliar a variabilidade genética de alevinos de piracanjuba (B. orbignyanus) de três meses de idade em relação a larvas de três dias e tentar averiguar possível presença de gargalos genéticos e mudanças na variabilidade ao longo do período de cultivo.
MATERIAL E MÉTODOS
A pesquisa foi realizada entre os meses de dezembro de 2014 e fevereiro de 2015, por meio de uma parceria entre a Universidade Estadual de Maringá (UEM) e a empresa AES-Tiête, localizada na cidade de Promissão, estado de São Paulo. As metodologias empregadas nesta pesquisa atenderam aos princípios éticos postulados pelo Conselho Nacional de Controle de Experimentação Animal, e foram aprovadas pela Comissão de Ética no Uso de Animais da Universidade Estadual de Londrina (Ceua∕UEL nº17156.2012.50).
Inicialmente, foram realizados acasalamentos de 12 reprodutores (nove machos e três fêmeas) de B. orbignyanus, oriundos da estação de hidrobiologia AES-Tietê, por meio do sistema seminatural de desova. Para a indução hormonal, foi seguida a metodologia descrita por Lopera-Barrero et al. (2014). As matrizes e os reprodutores foram alojados em um tanque circular, de aproximadamente 4m de diâmetro e 1,5m de profundidade, com sistema de circulação forçada de água, a fim de simular condições ambientais satisfatórias, onde houve a fertilização direta e aleatória pelos machos no momento da desova. A desova aconteceu após 160 horas∕grau. O escoamento da água do tanque ocorreu na porção central, o que permitiu a coleta dos ovos fertilizados e posterior transferência para a incubadora.
Os ovos foram incubados até a eclosão, e, após três dias, quando as larvas já haviam consumido a maior parte de sua reserva vitelínica, foram coletados 180 indivíduos de maneira aleatória, os quais foram eutanasiados por meio de choque térmico, pela imersão em água contendo gelo, com aproximadamente 1ºC. As amostras foram armazenadas em álcool 70% para posteriores análises genéticas.
Outros 1.000 indivíduos foram devidamente acondicionados em sacos plásticos com oxigênio e transportados para a Estação de Piscicultura UEM-Codapar, localizada no município de Maringá/PR. As larvas foram alojadas em dois tanques de alvenaria medindo 10m2 (5m x 2m) cada e cultivadas por três meses, até atingirem a fase de alevino. A qualidade de água durante esse período manteve-se dentro da faixa recomendada para produção de peixes, com valores de temperatura acima de 27ºC, oxigênio dissolvido acima de 6mg L-1 e pH entre 6,5 e 8,5. Ao final do período, foram coletadas aleatoriamente amostras de nadadeira caudal de 90 alevinos.
Foi realizada extração de DNA das larvas e da nadadeira caudal dos alevinos utilizando-se o protocolo descrito por Lopera-Barrero et al. (2008). Em seguida, o DNA foi quantificado com o uso de um espectrofotômetro PICODROP® (Picodrop Limited, Hinxton, United Kingdom) e padronizado na concentração final de 10ng µL-1. A integridade do DNA foi verificada em gel de agarose 1%, corado com SYBR Safe TM DNA Gel Stain (Invitrogen, Carlsbad, CA, USA), em eletroforese, conduzido com tampão TBE 0,5X (250mM Tris-HCL, 30mM ácido bórico e 41,5mM EDTA) durante uma hora a 70 volts. O gel foi revelado utilizando-se um transiluminador, sendo a imagem capturada pelo software L-PIX HE (Loccus Biotecnologia, São Paulo, Brasil).
Para as análises de variabilidade genética, foram utilizados cinco primers microssatélites heterólogos descritos por Sanches e Galetti (2006SANCHES, A.; GALETTI, P.M. Microsatellites loci isolated in the freshwater fish Brycon hilarii. Mol. Ecol. Notes, v.6, p.1045-1046, 2006. ) para Brycon hilarii (Bh5, Bh6, Bh8, Bh13 e Bh16). A amplificação do DNA foi realizada em termociclador Veriti® (Applied Biosystems®) para um volume final de reação de 15µL, utilizando-se 1X tampãoTris-HCL, 2,0mM MgCl2, 0,8µM de cada primer (forward e reverse), 0,4mM de cada dNTP, 1U Platinum Taq polymerase e 20ng de DNA. O DNA foi inicialmente desnaturado a 94ºC por quatro minutos e, então, submetido a 30 ciclos de desnaturação a 94ºC por 30 segundos, 30 segundos de anelamento (variando a temperatura conforme cada primer) e extensão a 72ºC por um minuto e, posteriormente uma extensão final a 72ºC por 10 minutos.
As amostras amplificadas foram submetidas à eletroforese em gel de poliacrilamida 10% e conduzidas em tampão durante sete horas. Para visualização dos alelos microssatélites, foi utilizada a coloração com nitrato de prata, e o gel foi fotografado com câmera Nikon CoolPix 5200. O tamanho dos alelos foi calculado utilizando-se DNA Ladder 100bp (Invitrogen, Carlsbad, CA, USA).
A frequência alélica, o número efetivo de alelos (Ne), a heterozigosidade observada (Ho) e a heterozigosidade esperada (He) foram calculados para cada locus utilizando-se o software GenAlex (Peakall e Smouse, 2012PEAKALL, R.; SMOUSE, P.E. GenALEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, v.28, p.2537-2539, 2012. ). A análise de variância molecular (AMOVA), o equilíbrio Hardy-Weinberg (Hw), o desequilíbrio e a diferenciação genética (Fst) entre as larvas e os alevinos foram obtidos mediante o uso do software Arlequim 3.0 (Excoffier e Lischer, 2010EXCOFFIER, L.; LISCHER, H.E.L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Resour., v.10, p.564-567, 2010. ). O índice de fixação (Fis) foi calculado utilizando-se o software FSTAT 2.9.3 (Goudet, 2005GOUDET, J. FSTAT: a program to estimate and test gene diversities and fixation indices (version 2.9.3.2), 2005. Available in: <http//www.unil.ch/izea/softwares/FSTat.html>. Accessed in: 12 Sep. 2017.
http//www.unil.ch/izea/softwares/FSTat.h...
). O nível de significância empregado para o Fst e Fis foi de 5% de probabilidade (P<0,05). A definição de Wright (1978WRIGHT, S. Evolution and genetics of population. Chicago: University of Chicago Press, 1978. 590p.) foi utilizada a fim de indicar diferenciação genética entre as populações (pequena, moderada, alta e elevada). A deficiência e o excesso de heterozigosidade foram obtidos utilizando-se o modelo de mutação infinita (IAM), o modelo de mutação stepwise (SMM) e o modelo de mutação em duas fases (TPM), por meio do uso do software Bottleneck (Cournet e Luikart, 1996COURNET, J.M.; LUIKART, G. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data. Genetics, v.144, p.2001-2014, 1996. ).
RESUTADOS E DISCUSSÃO
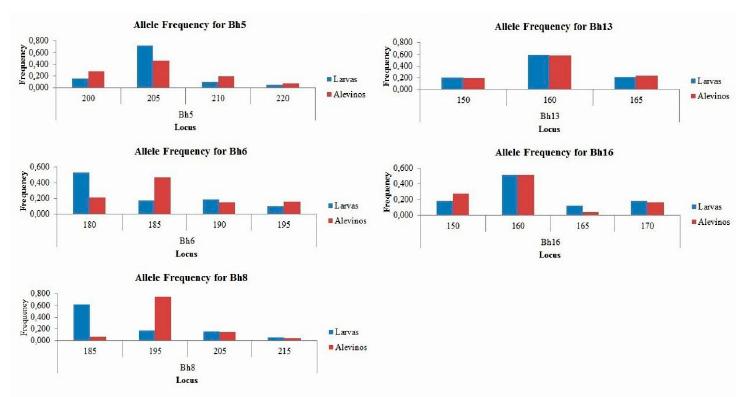
Os cinco loci produziram um total de 19 alelos. O tamanho dos alelos variou de 150pb (Bh13 e Bh16) a 220pb (Bh5) e a quantidade de alelos por locus variou de quatro (Bh5, Bh6, Bh8 e Bh16) a três (Bh13) (Fig. 1). O tamanho dos alelos foi semelhante ao encontrado por outros estudos com o gênero Brycon, como o Brycon hilari (Sanches e Galetti, 2006SANCHES, A.; GALETTI, P.M. Microsatellites loci isolated in the freshwater fish Brycon hilarii. Mol. Ecol. Notes, v.6, p.1045-1046, 2006. ; Bignardi et al., 2016BIGNARDI, A.B.; POVH, J.A.; ALVES, M.S. et al. Genetic variability of Brycon hilarii in a repopulation program. Braz. Arch. Biol. Technol., v.59, p.1-9, 2016. ) e B. orbignyanus (Carmo et al., 2015CARMO, F.M.S.; POLO, E.M.; SILVA, M.A. et al. Optimization of heterologous microsatellites in Piracanjuba. Pesqui. Agropecu. Bras., v.50, p.1236-1239, 2015. ), no entanto o número de alelos foi inferior no presente estudo. Não houve perda de alelos ao longo dos três meses de cultivo, ou seja, os mesmos alelos foram encontrados nas larvas e nos alevinos (ausência de alelos privados); não houve presença de alelos raros (frequência < 0,05). Em três loci (Bh5, Bh6, Bh13), os alelos de maior frequência foram mantidos nos dois grupos. O baixo número de alelos por locus (quatro a três alelos) pode estar relacionado à transferibilidade de primers desenvolvidos para a espécie Brycon hiilari para utilização em Brycon orbignyanus. Situação similar foi encontrada em estudo com B. orbignyanus com primers heterólogos desenvolvidos para Brycon opalinus (Rodriguez-Rodriguez et al., 2010).
A heterozigosidade observada (Ho) foi maior para as larvas (0,498) em relação aos alevinos (0,486). O número médio dos alelos efetivos, por outro lado, foi superior nos alevinos (2,413 e 2,589, para larvas e alevinos, respectivamente) (Tab. 1). Dados recentes na literatura mostram grande variação na Ho no estudo dessa espécie. Lopera-Barrero et al. (2014) encontraram valores que variaram de 0,823 a 0,945 para larvas obtidas do sistema de reprodução por extrusão e seminatural, respectivamente. Em populações naturais, valores entre 0,189 e 0,498 foram obtidos por Ashikaga et al. (2015ASHIKAGA, F.Y.; ORSI, M.L.; OLIVEIRA, M. et al. The endangered species Brycon orbignyanus: genetic analysis and definition of priority areas for conservation. Environ. Biol. Fish., v.98, p.1845-1855, 2015. ). Apesar da sutil diminuição apresentada na Ho dos alevinos em relação às larvas, a variabilidade genética apontada por esse índice pode ser considerada satisfatória para ambos os grupos (Ashikaga et al., 2015).
Número de alelos efetivos (Ne), heterozigosidade observada (Ho) e esperada (He), teste de probabilidade para o equilíbrio de Hardy-Weinberg (Hw), coeficiente de endogamia (Fis) e relação entre o número de locus com deficiência (Hd) e excesso (Hex) de heterozigotos calculados nas larvas e nos alevinos de B. orbignyanus
Os valores médios da heterozigosidade esperada (He) foram maiores que a média da heterozigosidade observada (Ho) tanto para as larvas quanto para os alevinos, indicando um déficit de heterozigotos. Esses resultados inferiram no coeficiente de endogamia (Fis), que apresentou resultados positivos e significativos (P>0,05) (Tab. 1). Houve desvio no equilíbrio de Hardy-Weinberg em quatro loci (Bh5, Bh6, Bh8 e Bh16) nas larvas e em quatro loci (Bh5, Bh6, Bh13 e Bh16) nos alevinos. Uma das principais explicações é a deriva genética, já que as flutuações aleatórias das frequências gênicas são mais acentuadas quando o número de indivíduos é reduzido (Caujapé-Castells, 2006), como é o caso de animais criados em pisciculturas. O desequilíbrio de ligação (P<0,05) entre os loci Bh6 com Bh13 e Bh6, e Bh13 com Bh16 nas larvas; e nos loci Bh5 com Bh6 e Bh13, e Bh8 com Bh13 e Bh6 nos alevinos fortalece essa hipótese.
Fatores como o efeito fundador (formação de nova população a partir de um número pequeno de reprodutores) e o efeito gargalo (redução drástica do tamanho populacional) podem implicar variação das frequências gênicas de maneira muito mais acentuada em populações mantidas em cativeiro (Caujapé-Castells, 2006; Frankham et al., 2008FRANKHAM, R.; BALLOU, J.D.; BRISCOE, D.A. (Eds.). Fundamentos de genética da conservação. Riberão Preto: Sociedade Brasileira de Genética, 2008. 262p. ). Nesse sentido, um número limitado de reprodutores existentes nesses programas formará uma progênie de milhares de indivíduos, o que favoreceria a perpetuação ou exclusão de certos alelos ao longo do tempo. De maneira análoga, a redução populacional devido à alta mortalidade nas primeiras fases de vida do B. orbignyanus pode favorecer o aparecimento de um gargalo genético. A análise de Bottleneck, por outro lado, demonstrou que o excesso de heterozigotos não foi significativo (P>0,05) para as larvas nos três modelos (IAM, SMM e TPM) e mostrou-se significativo (P<0,05) para os alevinos apenas para o modelo SMM.
Os resultados da AMOVA mostraram que essa variação genética (P<0,05) foi maior dentro dos grupos do que entre os grupos (Tab. 2). A diferenciação genética entre os estoques, segundo a classificação de Wright (1978WRIGHT, S. Evolution and genetics of population. Chicago: University of Chicago Press, 1978. 590p.), foi moderada. Em se tratando do mesmo lote de peixes, espera-se baixa diferenciação genética, contudo os resultados do presente estudo demonstraram que as diferentes fases de vida podem interferir nessa diferenciação.
Nas primeiras fases de vida, é comum que espécies como o dourado (Gomes et al., 2011GOMES, P.C.; RIBEIRO, R.P.; SIROL, R.N. et al. Diversidade genética de dourado utilizado em programas de repovoamento no rio Paranapanema. Pesqui. Agropecu. Bras., v.46, p.167-173, 2011. ) e a piracanjuba (Rodriguez-Rodriguez et al., 2010) sejam fortemente influenciadas por fatores ambientais, como a aclimatação no cativeiro, ou comportamentais, como o canibalismo, levando a altas taxas de mortalidade. Portanto, uma perda da variabilidade genética de um lote nos primeiros meses é comum devido a essas pressões seletivas (Gomes et al., 2011; Rodriguez-Rodriguez et al., 2010). Por essa razão, a aquisição de reprodutores com alta variabilidade genética e o correto manejo dos acasalamentos (Lopera-Barrero et al., 2015; Ribeiro et al., 2016RIBEIRO, R.P.; RODRIGUEZ-RODRIGUEZ, M.P.; RESENDE, E.K. et al. Genetic characteristics of Tambaqui broodstocks in the state of Rondônia, Brazil: implications on production and conservation. Semin. Cienc. Agrar., v.37, p.2375-2386, 2016. ) são fundamentais para a gestão da produção e conservação dessa espécie. Além disso, a soltura de animais por parte dos programas de repovoamento deve ser realizada com indivíduos que tenham maior perspectiva de sobrevivência, uma vez que a construção de barragens altera significativamente o regime hídrico natural e consequentemente a dinâmica dos estoques pesqueiros (Olden e Naiman, 2010OLDEN, J.D.; NAIMAN, R.J. Incorporating thermal regimes into environmental flows assessments: modifying dam operations to restore freshwater ecosystem integrity. Freshwater Biol., v.55, p.86-107, 2010.), principalmente nas fases iniciais de vida. Esse fato foi constatado por Casas-Mulet et al. (2016), os quais verificaram, por meio de modelagem matemática, que a probabilidade de mortalidade de salmão que habita rios represados é maior nos primeiros estágios de vida.
Segundo Gomes et al. (2011GOMES, P.C.; RIBEIRO, R.P.; SIROL, R.N. et al. Diversidade genética de dourado utilizado em programas de repovoamento no rio Paranapanema. Pesqui. Agropecu. Bras., v.46, p.167-173, 2011. ), a avaliação da diversidade genética nos dois estágios de vida (larvas e alevinos) é importante para direcionar as atividades dentro de um programa de repovoamento, já que, por meio das análises das larvas, pode-se inferir as características genéticas que foram obtidas na nova geração e, mediante as análises dos alevinos, pode-se estimar a variabilidade dos animais que serão soltos no ambiente. No entanto, de acordo com os autores, normalmente apenas a análise genética das larvas é realizada como índice de diversidade genética. Apesar da sutil diferença na variabilidade genética dos dois grupos encontrada no presente trabalho, recomenda-se a avaliação nos dois estágios de vida pelos motivos já citados. É possível que a pressão seletiva enfrentada durante o desenvolvimento desses animais não tenha sido suficiente para promover uma drástica queda na variabilidade, entretanto a análise do efeito gargalo demonstrou um excesso de heterozigotos significativo por meio do modelo SMM, o que caracteriza um efeito gargalo recente.
Por fim, este estudo encontrou que alevinos que serão liberados no ambiente possuem adequada variabilidade genética. Todavia, devido ao fato de o coeficiente de endogamia ser positivo, significativo, apresentar aumento durante as fases de vida e, ainda, com possibilidade de gargalo genético, fica evidente a necessidade de futuras análises genéticas nas próximas gerações de larvas e alevinos. Mais estudos serão necessários para avaliar a variabilidade genética dos reprodutores e das matrizes nos programas de conservação, bem como o estado genético das populações naturais. Acredita-se que, dessa forma, será possível conduzir os manejos reprodutivos nas centrais de repovoamento sem que haja diminuição da diversidade genética nos alevinos que serão liberados no ambiente.
CONCLUSÃO
Os estoques de larvas e alevinos de piracanjuba demonstraram adequada variabilidade genética. No entanto, os valores positivos no coeficiente de endogamia e a existência de gargalo genético na fase de alevino atentam para o risco de declínio de variabilidade genética e reforçam a necessidade de constantes monitoramentos genéticos antes da liberação desses indivíduos no ambiente natural.
REFERÊNCIAS
- AGOSTINHO, A.A.; GOMES, L.C.; PELICICE,F.M (Eds.). Ecologia e manejo de recursos pesqueiros em reservatórios do Brasil. Maringá: Eduem, 2007. 501p.
- APRAHAMIAN, M.; SMITH, K.M.; McGINNITY, P. et al. Restocking of salmonids-opportunities and limitations. Fish. Res., v.62, p.211-227, 2003.
- ASHIKAGA, F.Y.; ORSI, M.L.; OLIVEIRA, M. et al. The endangered species Brycon orbignyanus: genetic analysis and definition of priority areas for conservation. Environ. Biol. Fish., v.98, p.1845-1855, 2015.
- BIGNARDI, A.B.; POVH, J.A.; ALVES, M.S. et al. Genetic variability of Brycon hilarii in a repopulation program. Braz. Arch. Biol. Technol., v.59, p.1-9, 2016.
- BORBA, M.R.; FRACALOSSI, D.M.; PEZZATO, L.E. Dietary energy requirement of piracanjuba fingerlings, Brycon orbignyanus, and relative utilization of dietary carbohydrate and lipid. Aquacult. Nutr., v.12, p.183-191, 2006.
- CARMO, F.M.S.; POLO, E.M.; SILVA, M.A. et al. Optimization of heterologous microsatellites in Piracanjuba. Pesqui. Agropecu. Bras., v.50, p.1236-1239, 2015.
- CAUJAPÉ-CASTELLS, J. (Ed.). Brújula para botánicos desorientados en la genética de poblaciones. Espanha: Exegen, 2006. 133p.
- CASAS-MULET, R.; SALTVEIT, S.J.; ALFREDSEN, K.T. Hydrological and thermal effects of hydropeaking on early life stages of salmonids: a modelling approach for implementing mitigation strategies. Sci. Total Environ., v.573, p.1660-1672, 2016.
- COURNET, J.M.; LUIKART, G. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data. Genetics, v.144, p.2001-2014, 1996.
- EXCOFFIER, L.; LISCHER, H.E.L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Resour., v.10, p.564-567, 2010.
- FRANKHAM, R.; BALLOU, J.D.; BRISCOE, D.A. (Eds.). Fundamentos de genética da conservação. Riberão Preto: Sociedade Brasileira de Genética, 2008. 262p.
- GOMES, P.C.; RIBEIRO, R.P.; SIROL, R.N. et al. Diversidade genética de dourado utilizado em programas de repovoamento no rio Paranapanema. Pesqui. Agropecu. Bras., v.46, p.167-173, 2011.
- GONZÁLEZ-WANGÜEMERT, M.; FERNÁNDEZ, T.V.; PÉREZ-RUZAFA, A. et al. Genetic considerations on the introduction of farmed fish in marine protected areas: the case of study of white seabream restocking in the Gulf of Castellammare (Southern Tyrrhenian Sea). J. Sea Res., v.68, p.41-48, 2012.
- GOUDET, J. FSTAT: a program to estimate and test gene diversities and fixation indices (version 2.9.3.2), 2005. Available in: <http//www.unil.ch/izea/softwares/FSTat.html>. Accessed in: 12 Sep. 2017.
» http//www.unil.ch/izea/softwares/FSTat.html - LOPERA-BARRERO, N.M.; ALVAREZ, C.; RODRIGUEZ-RODRIGUEZ, M. et al. Diversidade genética e paternidade de progênies de Brycon orbignyanus obtidas por diferentes sistemas reprodutivos. Semin. Cienc. Agrar., v.35, p.541, 2014.
- LOPERA-BARRERO, N.M.; POVH, J.A.; SIROL, R.N. et al. Genetic diversity of pacu and piapara broodstocks in restocking programs in the rivers Paraná and Paranapanema (Brazil). Semin. Cienc. Agrar., v.37, p.2365, 2016.
- LOPERA-BARRERO, N.M.; RIBEIRO, R.P.; SIROL R.N. et al. Caracterización genética de lotes de Brycon orbignyanus utilizados em programas de repoblamiento. Rev. MVZ Córdoba, v.13, p.1110-1119, 2008.
- LOPERA-BARRERO, N.M.; RODRIGUEZ-RODRIGUEZ, M.P.; FORNARI, D.C. et al. Genetic variability of broodstocks of Tambaqui (Teleostei - characidae) from the northeast region of Brazil. Semin. Cienc. Agrar., v.36, p.4013, 2015.
- MACHADO, A.B.M.; DRUMMOND, G.M.; PAGLIA, A.P. (Eds.). Livro vermelho da fauna brasileira ameaçada de extinção. Brasília: MMA, 2008. 1420p.
- MARIA, A.N.; VIVEIROS, A.T.M.; FREITAS, R.T.F. et al. Extenders and cryoprotectants for cooling and freezing of piracanjuba (Brycon orbignyanus) semen, an endangered Brazilian teleost fish. Aquaculture, v.260, p.298-306, 2006.
- MURGAS, L.D.S.; MILIORINI, A.B.; FRANCISCATTO, R.T. et al. Viabilidade espermática do sêmen de piracanjuba (Brycon orbignyanus) resfriado a 4°C. Rev. Bras. Zootec., v.33, p.1361-1365, 2004.
- OLDEN, J.D.; NAIMAN, R.J. Incorporating thermal regimes into environmental flows assessments: modifying dam operations to restore freshwater ecosystem integrity. Freshwater Biol., v.55, p.86-107, 2010.
- PANARARI-ANTUNES, R.S.; PRIOLI, A.J.; PIOLI, S.M.A.P. et al. Genetic variability of Brycon orbignyanus (valenciennes, 1850) (characiformes: characidae) in cultivated and natural populations of the upper paraná river, and implications for the conservation of the species. Braz. Arch. Biol. Technol., v.54, p.839-848, 2011.
- PEAKALL, R.; SMOUSE, P.E. GenALEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, v.28, p.2537-2539, 2012.
- RIBEIRO, R.P.; RODRIGUEZ-RODRIGUEZ, M.P.; RESENDE, E.K. et al. Genetic characteristics of Tambaqui broodstocks in the state of Rondônia, Brazil: implications on production and conservation. Semin. Cienc. Agrar., v.37, p.2375-2386, 2016.
- RODRIGUEZ-RODRIGUEZ, M.P.; LOPERA-BARRERO, N.M.; RIBEIRO, R.P. et al. Diversidad genética de piracanjuba usada en programas de repoblación con marcadores microsatélites. Pesqui. Agropecu. Bras., v.45, p.56-63, 2010.
- SANCHES, A.; GALETTI, P.M. Microsatellites loci isolated in the freshwater fish Brycon hilarii. Mol. Ecol. Notes, v.6, p.1045-1046, 2006.
- WRIGHT, S. Evolution and genetics of population. Chicago: University of Chicago Press, 1978. 590p.
Datas de Publicação
-
Publicação nesta coleção
06 Jun 2019 -
Data do Fascículo
Mar-Apr 2019
Histórico
-
Recebido
26 Set 2017 -
Aceito
02 Jul 2018