Resumos
Dados de 769.925 animais da raça Nelore, nascidos de 1965 a 2000, em 30 regiões distintas do Brasil, foram utilizados como base de dados para desenvolvimento de um programa computacional flexível, de fácil uso, que permitisse a identificação de linhagens fundadoras de rebanhos, com filtro por país, estado, região ou rebanho. Foi escolhida a região Centro-Oeste, representativa do Brasil, para o estudo pormenorizado do efeito citoplasmático sobre característica de peso aos 205 e 365 dias de idade de animais Nelore. Os componentes de variância de linhagem citoplasmática foram iguais a 0,58x10-3 para peso aos 205 dias e 0,80x10-3 para o peso aos 365 dias e contribuíram com 1,00x10-5% e 9,90x10-6% da variância fenotípica total. Os componentes de variância aditivo direto foram responsáveis por 11 e 21% da variação fenotípica total dos pesos aos 205 e 365 dias de idade.
bovino de corte; linhagem citoplasmática; informática
Records on 769,925 Nelore animals born from 1965 to 2000, in 30 regions of Brazil were used as base data to develop a flexible a ease use computational program for identifying maternal base lines in herds, allowing filter by country, state, region and herd. The Center-West region was selected for a detailed study of cytoplasmatic lines on weaning and yearling weights of Nelore animals. The estimated variance components for citoplasmatic lines were .58 x 10-3 and .80 x 10-.8 for weaning and yearling weights, respectively, and accounted for 1.00x10-5 and 9.90x10-6 of total weights variance, respectively. The direct additive component accounts for 11 and 12% of the total phenotypic variance of weaning and yearling weights.
beef cattle; citoplasmatic line; computer science
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Desenvolvimento do software DRLinhagem para detecção de linhagens citoplasmáticas e avaliação de seu efeito sobre os pesos à desmama e ao ano de idade de animais Nelore no Centro-Oeste brasileiro
Development of a DRLinhagem software to detect citoplasmatic lines and their effects on weaning and yearling weights of Nelore animals from the center west part of Brazil
R.V. VenturaI; M.A. SilvaII, IV; N.L. DionelloIII; D.F.D. MagalhãesI; J.C.C. PereiraII, IV; V.M. PennaII; J.A.G. BergmannII, IV; F.E. MadalenaII, IV
IAluno de pós-graduação - EV-UFMG Belo Horizonte, MG
IIEscola de Veterinária - UFMG Caixa Posta 567 30123-970 Belo Horizonte, MG
IIIDepartamento de Zootecnia - FAEM-UFPel Pelotas, RS
IVBolsista do CNPq
RESUMO
Dados de 769.925 animais da raça Nelore, nascidos de 1965 a 2000, em 30 regiões distintas do Brasil, foram utilizados como base de dados para desenvolvimento de um programa computacional flexível, de fácil uso, que permitisse a identificação de linhagens fundadoras de rebanhos, com filtro por país, estado, região ou rebanho. Foi escolhida a região Centro-Oeste, representativa do Brasil, para o estudo pormenorizado do efeito citoplasmático sobre característica de peso aos 205 e 365 dias de idade de animais Nelore. Os componentes de variância de linhagem citoplasmática foram iguais a 0,58x10-3 para peso aos 205 dias e 0,80x10-3 para o peso aos 365 dias e contribuíram com 1,00x10-5% e 9,90x10-6% da variância fenotípica total. Os componentes de variância aditivo direto foram responsáveis por 11 e 21% da variação fenotípica total dos pesos aos 205 e 365 dias de idade.
Palavras-chave: bovino de corte; linhagem citoplasmática; informática
ABSTRACT
Records on 769,925 Nelore animals born from 1965 to 2000, in 30 regions of Brazil were used as base data to develop a flexible a ease use computational program for identifying maternal base lines in herds, allowing filter by country, state, region and herd. The Center-West region was selected for a detailed study of cytoplasmatic lines on weaning and yearling weights of Nelore animals. The estimated variance components for citoplasmatic lines were .58 x 10-3 and .80 x 10-.8 for weaning and yearling weights, respectively, and accounted for 1.00x10-5 and 9.90x10-6 of total weights variance, respectively. The direct additive component accounts for 11 and 12% of the total phenotypic variance of weaning and yearling weights.
Keywords: beef cattle; citoplasmatic line; computer science
INTRODUÇÃO
No mercado brasileiro existem disponíveis diversos programas para computador desenvolvidos para controle e manejo de rebanhos, todos eles com características próprias, mas, que exigem treinamento intenso dos usuários para minimização de perdas do produtor (Lopes et al., 2007).
Um software de qualidade deve oferecer ao usuário toda a informação requisitada, de maneira eficaz e confiável. Segundo Bergmann (1998), números extraídos de fichários zootécnicos e arquivos de registro animal são de pouca utilidade e não se tornam dados até que sejam organizados sistematicamente e colocados numa forma que permita sua utilização e manipulação.
É interessante verificar que mesmo com boa coleta e montagem da base de dados, as informações surgem à medida que esses dados são manipulados, gerando-se resultados na forma de relatórios e arquivos de saída. Entretanto, deve-se ressaltar que é de extrema importância que dados com qualidade e idoneidade sejam fontes para arquivos de leitura em qualquer software.
No passado, componentes citoplasmáticos eram ignorados e desconsiderados nas estimativas dos parâmetros genéticos das características produtivas dos diversos rebanhos. No entanto, por intermédio de genética molecular, pesquisas recentes demonstram que o citoplasma e organelas extra-nucleares, incluindo mitocôndrias que contém mtDNA (DNA mitocondrial) são herdadas da fêmea, em mamíferos. Isto é a base da herança uniparental ou materna, em que a prole sempre se assemelha ao genitor feminino.
Dessa forma, a herança citoplasmática pode ser um dos fatores que causam diferença entre animais e rebanhos. Isso se deve ao fato do gameta feminino contribuir com grande parte do citoplasma do zigoto e o gameta masculino com o DNA do núcleo. Diferenças entre cruzamentos recíprocos sugerem também envolvimento citoplasmático.
Mitocôndria são organelas auto-replicáveis localizadas no citoplasma da maioria das células vivas, exceto bactérias, cianobactérias e eritrócitos maduros. Segundo Boettcher et al. (1996), as mitocôndrias são estruturas complexas responsáveis por aproximadamente 90% da energia celular consumida por um organismo mamífero. As mitocôndrias são responsáveis pelo processo da forforilação oxidativa (síntese de ATP a partir da energia liberada no transporte de elétrons na cadeia respiratória.). Dessa forma, pode-se esperar que modificações no genoma mitocondrial tenham reflexos sobre o metabolismo celular, e em conseqüência sobre o desempenho produtivo dos animais.
A herança citoplasmática pode ser avaliada a partir das informações de pedigree dos animais, percorrendo por intermédio da linha materna todos os registros de produção das fêmeas até as fundadoras do rebanho.
O mecanismo de busca de linhagens fundadoras é realizado com auxilio de técnicas computacionais, com desenvolvimento de softwares eficientes para a manipulação dos arquivos de registros de animais, em razão do tamanho do arquivo da linhagem fundadora.
O objetivo deste estudo foi desenvolver um programa computacional flexível, de fácil uso para identificação de linhagens fundadoras de rebanhos, permitindo-se estudo (filtro) por país, estado, região ou rebanho, e utilizando informações de animais da raça Nelore da região Centro-Oeste para o estudo pormenorizado do efeito citoplasmático sobre os pesos à desmama e a um ano de idade.
MATERIAL E MÉTODOS
O dados utilizados são provenientes de registros de animais da Associação Brasileira de criadores da raça Zebu (ABCZ), cedidos pela Embrapa Gado de Corte (Campo Grande, MS). O banco de dados contém registros de 769.925 animais nascidos de 1965 a 2000, em 30 regiões distintas.
As características estudadas foram os pesos aos 205 dias (P205) e peso aos 365 dias (P365). A verificação de consistência dos dados foi feita por meio do software SAS em busca de dados que não condiziam com a realidade abordada em outros estudos, o que reduziu o número de informações de 769.925 para 233.670 animais, após o descarte de dados.
Os animais foram inicialmente agrupados em quatro regiões - Centro-Oeste, Sudeste, Sul e Nordeste - de acordo com a freqüência de animais obtida em cada região. Após a eliminação de áreas com freqüência inferior a 3%, nova configuração dos dados nas quatro regiões ficou assim estabelecida: 1 região Sudeste, envolvendo Minas Gerais e São Paulo; 2 região Centro-Oeste, envolvendo Mato Grosso, Mato Grosso do Sul e Goiás; 3 região Nordeste, formada pelo estado da Bahia e 4 região Sul, formada pelo estado do Paraná.
A linhagem materna foi obtida por meio do software DRLinhagem, desenvolvido especialmente para este estudo, utilizando-se da tecnologia Java. Sua obtenção caracteriza-se pelo reconhecimento e detecção das fêmeas fundadoras de cada rebanho, obtidas por informações dos animais até as primeiras fêmeas que originaram os animais subseqüentes.
Esse software permite a inclusão de novas restrições aos dados, tais como: 1 - filtro de dados e obtenção de linhagens por país; 2 - filtro de dados e obtenção de linhagens por estado; 3 - filtro de dados e obtenção de linhagens por região; 4 - filtro de dados e obtenção de linhagens por rebanho e 5 - número mínimo de gerações a ser considerada para obtenção da linhagem fundadora.
Para a detecção das linhagens fundadoras de rebanho, usou-se do recurso List ou lista, disponível na tecnologia Java. A metodologia adotada pode ser assim descrita: 1 - existência de um arquivo de entrada contendo dados de mãe e filha correspondente; 2 - criação de três novas listas denominadas listaMae, listaFilha e listaLinhagem, respectivamente, com a função de armazenar dados da mãe, filha e a última lista responsável pela armazenagem das linhagens fundadoras; 3 - leitura dos dados contidos no arquivo de entrada e alocação dinâmica nas listas recém criadas e 4 - pesquisa do registro da mãe em listaFilha e pesquisa do registro da filha em listaLinhagem, em uma mesma instrução ou comando. Por meio de uma busca, caso a mãe seja localizada em listaFilha, não é promovida a inserção dessa mãe na listaLinhagem. Caso a filha esteja em listaLinhagem faz-se a remoção dessa filha da listaLinhagem.
Para fins de entendimento e simulação utilizou-se de pequena massa de dados, com demonstração do uso e criação de listas, apresentadas na diagrama 1.
Foram utilizados dois modelos matemáticos para estimar componentes de variâncias e parâmetros genéticos dos pesos aos 205 dias de idade - p205 (peso à desmama) - e peso aos 365 dias de idade (peso a um ano de idade) - p365 -, com inclusão ou não do efeito citoplasmático ao modelo de análise. Análises univariadas foram desenvolvidas com base nos modelos propostos como se segue: Modelo 1 - efeitos fixos de grupo contemporâneo, geração em que se encontra o animal, sexo, rebanho, idade e efeitos aleatórios genético aditivo direto e aditivo materno; Modelo 2 - efeitos fixos de grupo contemporâneo, geração em que se encontra o animal, sexo, rebanho, idade e efeitos aleatórios genético aditivo direto e aditivo materno, e inclusão do efeito aleatório de linhagem citoplasmática.
O modelo estatístico mais completo, em forma matricial, para análise dos dados foi:
y = Xb + Zg + Mm +Eec + e , em que:
y = vetor de observações X, Z, M, E = matrizes de incidência destinada à associação dos efeitos ao vetor de observações;
b = vetor dos efeitos fixos de grupo contemporâneo, geração, sexo, rebanho e idade;
g = vetor do efeito genético aditivo direto;
m = vetor do efeito genético aditivo materno;
ec = vetor do efeito de linhagem citoplasmática;
e = vetor dos efeitos residuais.
Adotaram-se as seguintes premissas no modelo:
E[y] = Xb, E[g]= 0, E[m]= 0, E[ec]= 0, E[e]= 0, Var[g]= As2g, Var[m]= As2m, Cov[g,m]= Asgm, Var[ec]= ICLMs2ec e Var[e]= IRegs2e. A é conhecida como matriz de relacionamento genético e I, a matriz identidade.
Os componentes de variância e herdabilidades foram estimados pelo Método de Maxima Verossimilhança Restrita (REML), usando o algoritmo Derivative Free Restricted Maximum Likelihood (DFREML), descrito por Meyer (1993), e apresentado por Silva et al. (2006).
RESULTADOS E DISCUSSÃO
Para a execução do software DRLinhagem, tendo como fonte de entrada os arquivos criados para o Brasil e as regiões Sudeste, Centro-Oeste, Sul e Nordeste, separadamente, o software exige que seja informado um número mínimo de gerações de contribuição como filtro de dados. O número total de fêmeas e de linhagens fundadoras, que contribuíram por uma a sete gerações (filtro de dados), em todo o Brasil e nas quatro regiões estudadas, é apresentado nas Tab. 1 e 2, respectivamente.
Após filtragem dos dados de acordo com o número de mínimo de gerações e do número total de fêmeas (Tab. 1), apenas parte deles foi identificado como fêmeas fundadoras (Tab. 2), sendo as remanescentes fêmeas descendentes diretas das linhagens fundadoras (linhagens citoplasmáticas).
O número de fêmeas fundadoras de rebanhos nas regiões brasileiras, cotejado com as informações de todo o país, dentro de cada filtro de dados (número gerações de contribuição) foi maior nas regiões Sudeste e Centro-Oeste por disporem de maior número de animais e, possivelmente, por apresentarem melhor controle nas anotações de produção e reprodução dos animais. Na certa, o número de contribuições encontrado no Brasil todo é baixo, mas poderia ser muito maior caso o registro das informações fosse realizado com mais rigor.
A contribuição das linhagens citoplasmáticas apresentadas na Tab. 2 para as gerações sucessivas, dentro de cada filtro de contribuição de gerações, para todas as regiões, é apresentada, em percentagem, nas Tab. 3 a 6.
De modo geral, as regiões Sudeste e Centro-Oeste apresentaram porcentagens semelhantes dentro de cada filtro de dados na geração base e nas gerações sucessivas. Assim, as percentagens observadas para as duas regiões foram, respectivamente, 38,3% e 37,6% para fêmeas fundadoras de rebanho quando o filtro de dados adotado foi de uma geração como contribuição mínima. O mesmo comportamento foi observado para os demais valores de contribuição ao longo das gerações nas diversas regiões, podendo ser destacada a semelhança encontrada nos dados da geração 4, cujas percentagens foram 0,01; 0,09; 0,76; e 11,1% para a região Sudeste e 0,02; 0,10; 0,79; e 9,7% para a região Centro-Oeste.
É possível que a maior disponibilidade de dados e o maior rigor nas anotações, sejam responsáveis pelo maior número e percentagens de fêmeas descendentes nas gerações 1, 2, 3 e 4 observados na região Centro-Oeste.
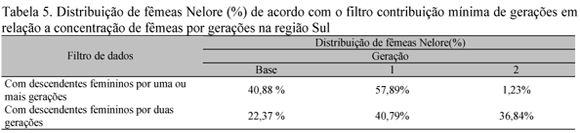
As regiões Sul e Nordeste apresentam menor número de fêmeas que contribuíram para a continuidade das linhagens citoplasmáticas, não ultrapassando três gerações de contribuição. A região Sul apresentou menor valor para contribuição de gerações apesar de apresentar número maior de animais na geração base para filtro mínimo de gerações igual a 1, quando comparada à região Nordeste. Os números de fêmeas fundadoras encontradas para esse filtro de dados foram de 930 (40,9%) e 905(35,1%) animais, respectivamente, nas regiões Sul e Nordeste. Para a região Sul não foram encontradas fêmeas que contribuíram com mais de duas gerações.
As estimativas dos componentes de variâncias do peso aos 205 e 365 dias de idade da região Centro-Oeste são apresentadas nas Tab. 7 e 8, utilizando-se dois modelos distintos. Conforme visto, a diferença na elaboração dos dois modelos se deu exclusivamente pela inclusão da linhagem citoplasmática como efeito aleatório no segundo modelo.
De acordo com os resultados apresentados nas Tab. 7 e 8, as estimativas dos componentes de variância fenotípica total (s2p), residual (s2e), aditivo direto (s2a) e aditivo materno (s2m) não foram diferentes nas análises dos dois modelos, para as duas características avaliadas, o que demonstra pouca influência da linhagem materna sobre as características avaliadas. As estimativas dos componentes de variância de linhagem citoplasmática foram iguais a 0,58x10-3 para peso aos 205 dias e 0,80x10-3 para o peso aos 365 dias (Tab. 7 e 8) e contribuíram com 1,00x10-5% e 9,90x10-6% da variância fenotípica total. Por outro lado, as estimativas dos componentes de variância aditivo direto foram responsáveis por 11 e 21% da variação fenotípica total dos pesos aos 205 e 365 dias de idade, que representam as estimativas de herdabilidade das duas características.
No Brasil, Alencar et al. (1997) também estudaram a influência da linhagem citoplasmática sobre peso ao nascimento (PN), à desmama (PD) e peso aos 365 dias (PA) em bovinos da raça Canchim. Os autores admitiram que as vacas da raça Indubrasil do primeiro acasalamento Charolês x Indubrasil se constituíram em linhagens citoplasmáticas, considerando esse efeito como aleatório. As estimativas dos componentes de variância foram iguais a 0,45; 0,59 e 0,00 para o PN, PD e PA respectivamente, sendo responsáveis por 1,3; 0,05 e 0,00% da variância fenotípica total. Dessa forma, os autores concluem que a linhagem citoplasmática não apresentou efeitos significativos sobre características de crescimento em animais da raça Canchim.
Pelicioni (2001), ao utilizar o procedimento de máxima verossimilhança restrita livre de derivada, estudaram efeitos da linhagem materna sobre o peso ao nascer (PN) e ganho de peso diário (GPD) na pré-desmama em bovinos da raça Caracu. Por intermédio de seis diferentes modelos concluem os autores que a variância atribuída à linhagem citoplasmática não explicou significativamente as variações fenotipicas dessas características.
As características de desenvolvimento em animais da raça Nelore foram analisadas por Gunski (2001), por meio de três modelos distintos pela inserção da linhagem citoplasmática como efeito fixo ou aleatório, ou por desconsiderar este efeito. Os resultados indicaram que estimativas de herdabilidade do peso aos 120 dias variaram de 0,22 a 0,24 (direta) e de 0,13 a 0,19 (materna). Portanto, a inserção do mtDNA como variável fixa ou aleatória nas análises não influencia os parâmetros genéticos estimados das características de crescimento pré e pós-desmama na raça Nelore.
Em razão da baixíssima magnitude dos valores, nenhum teste estatístico foi realizado para avaliar o efeito dos componentes de linhagem citoplasmática para peso aos 205 e 365 dias de idade.
CONCLUSÕES
Foi desenvolvido o software DRLinhagem de alta eficiência e flexibilidade que permite filtros por país, região, estado e por rebanho, para fins de estudos de herança citoplasmática. O software utiliza como entrada de dados qualquer arquivo tipo texto, com dados separados por espaços ou tabulações. O arquivo de saída fornece de forma individualizada o número da linhagem citoplasmática e a geração a que pertence o animal. A herança citoplasmática não é importante para explicar variações de peso aos 205 e 365 dias de idade de animais Nelore.
Recebido em 1 de agosto de 2006
Aceito em 8 de outubro de 2007
E-mail: rvventura@gmail.com
- ALENCAR, M.M.; TREMATORE, R.L.; BARBOSA, P.F. et al. Estudo da influencia da linhagem citoplasmática sobre pesos em bovinos da Raça Canchim. In: REUNIÃO ANUAL DA SOCIEDADE BRASILEIRA DE ZOOTECNIA, 34., 1997, Juiz de Fora. Anais ... Juiz de Fora: SBZ, 1997. p.230-232.
- BERGMANN, J.A.G. Dos números ao conhecimento e à aplicação, passando pela estatística. In: SIMPÓSIO NACIONAL SOCIEDADE BRASILEIRA DE MELHORAMENTO ANIMAL, 2., 1998, Uberaba. Anais... Uberaba: SBMA, 1998. p.247-251.
- BOETTCHER, P.J.; KUHN, M.T.; FREEMAN, A.E. Impacts of cytoplasmic inheritance on genetic evaluations. J. Dairy Sci., v.79, p. 663-675, 1996.
- GUNSKI, R.J. Efeito do DNA mitocondrial sobre características de crescimento na raça Nelore 2001. 69f. Tese (Doutorado) Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, SP.
- LOPES, M.A.; LAGO, A.A.; CÓCARO, H. Uso de softwares para gerenciamento de rebanhos bovinos leiteiros. Arq. Bras. Med. Vet. Zootec., v.59, p.547-549, 2007.
- MEYER, K. DFREML. User notes, version 2.1. Armidale: University of New England, 1993.
- PELICIONI, L. C. Efeito da linhagem citoplasmática sobre o peso ao nascer e o ganho médio diário na pré-desmama em bovinos da raça Caracu. Rev. Bras. Zootec., v.30, p. 101-108, 2001.
- SILVA, M.A.; SARMENTO, J.L.R.; TORRES, R.A. et al. Manual de utilização do programa DFREML. Belo Horizonte: FEPMVZ, 2006. 103p.
- SUN MICROSYSTEMS. Disponível em: <http://www.java.sun.com/>. Acessado em: 12 jan. 2006.
» link
Datas de Publicação
-
Publicação nesta coleção
02 Jan 2008 -
Data do Fascículo
Out 2007
Histórico
-
Aceito
08 Out 2007 -
Recebido
01 Ago 2006