Resumos
O objetivo deste trabalho foi caracterizar a variabilidade genética nos parentais (G0) e em três gerações consecutivas (G1, G2 e G3) de tilápia Gift (genetically improved farmed tilapia), por meio de marcadores microssatélites. Trezentos e sessenta indivíduos, provenientes do programa de melhoramento da Universidade Estadual de Maringá, foram selecionados quanto ao ganho de peso. O total de 21 alelos foi encontrado nos cinco loci microssatélites polimórficos (G12292, UNH140; G12311, UNH159; G12312, UNH160; G12314, UNH162; e G12315, UNH163), com número médio entre três e cinco alelos por locus. As frequências alélicas variaram de 0,017 (UNH160 - G2) a 0,750 (UNH160 - G0). A heterozigosidade média observada foi de 0,501, 0,391, 0,531 e 0,503 para G0, G1, G2 e G3, respectivamente. O coeficiente de endogamia médio foi 0,192 (G0), 0,401 (G1), 0,230 (G2) e 0,301 (G3). Todas as gerações apresentaram desvio no equilíbrio de Hardy-Weinberg, com desequilíbrio de ligação na maioria dos loci. Exceto para a G1, a heterozigosidade foi mantida nas gerações G2 e G3, o que indica que não há perda significativa de variabilidade genética no programa de melhoramento.
Oreochromis niloticus; coeficiente de endogamia; frequência alélica; tilapicultura; variabilidade genética
The objective of this work was to characterize the genetic variability in parental (G0) and three subsequent generations (G1, G2, and G3) of genetically improved farmed tilapia (Gift) using microsatellite markers. Three hundred sixty individuals from the breeding program of Universidade Estadual de Maringá, PR, Brazil, were selected for weight gain. A total of 21 alleles was found in all five polymorphic microsatellite loci (G12292, UNH140; G12311, UNH159; G12312, UNH160; G12314, UNH162; and G12315, UNH163), with an average number between three and five alleles per locus. Allele frequencies ranged from 0.017 (UNH160 - G2) to 0.750 (UNH160 - G0). The average observed heterozygosity was 0.501, 0.391, 0.531, and 0.503 for G0, G1, G2, and G3, respectively. The average inbreeding coefficient was 0.192 (G0), 0.401 (G1), 0.230 (G2), and 0.301 (G3). All generations showed deviation from Hardy-Weinberg equilibrium, with linkage disequilibrium in most loci. Except for G1, the heterozygosity was maintained in the G2 and G3 generations, which indicates that there is no significant loss of genetic variability in the breeding program.
Oreochromis niloticus; inbreeding coefficient; allele frequency; tilapia farming; genetic variability
PISCICULTURA
Caracterização genética de gerações de tilápia Gift por meio de marcadores microssatélites
Genetic characterization of Gift tilapia generations using microsatellite markers
Maria Del Pilar Rodriguez-RodriguezI; Nelson Mauricio Lopera-BarreroII; Lauro VargasI; Daniele Menezes AlbuquerqueI; Elenice Souza dos Reis GoesI; Odimari Pricila Pires do PradoII; Ricardo Pereira RibeiroI
IUniversidade Estadual de Maringá, Avenida Colombo, nº 5.790, CEP 87020-900 Maringá, PR. E-mail: rodrigpilar@gmail.com, lvargas@uem.br, rpribeiro@uem.br, danielemenezes2003@yahoo.com.br, elenicesreis@yahoo.com.br
IIUniversidade Estadual de Londrina, Rodovia Celso Garcia Cid, PR-445, Km 380, CEP 86051-980 Londrina, PR. E-mail: nmlopera@uel.br, odimari@uel.br
RESUMO
O objetivo deste trabalho foi caracterizar a variabilidade genética nos parentais (G0) e em três gerações consecutivas (G1, G2 e G3) de tilápia Gift (genetically improved farmed tilapia), por meio de marcadores microssatélites. Trezentos e sessenta indivíduos, provenientes do programa de melhoramento da Universidade Estadual de Maringá, foram selecionados quanto ao ganho de peso. O total de 21 alelos foi encontrado nos cinco loci microssatélites polimórficos (G12292, UNH140; G12311, UNH159; G12312, UNH160; G12314, UNH162; e G12315, UNH163), com número médio entre três e cinco alelos por locus. As frequências alélicas variaram de 0,017 (UNH160 - G2) a 0,750 (UNH160 - G0). A heterozigosidade média observada foi de 0,501, 0,391, 0,531 e 0,503 para G0, G1, G2 e G3, respectivamente. O coeficiente de endogamia médio foi 0,192 (G0), 0,401 (G1), 0,230 (G2) e 0,301 (G3). Todas as gerações apresentaram desvio no equilíbrio de Hardy-Weinberg, com desequilíbrio de ligação na maioria dos loci. Exceto para a G1, a heterozigosidade foi mantida nas gerações G2 e G3, o que indica que não há perda significativa de variabilidade genética no programa de melhoramento.
Termos para indexação:Oreochromis niloticus, coeficiente de endogamia, frequência alélica, tilapicultura, variabilidade genética.
ABSTRACT
The objective of this work was to characterize the genetic variability in parental (G0) and three subsequent generations (G1, G2, and G3) of genetically improved farmed tilapia (Gift) using microsatellite markers. Three hundred sixty individuals from the breeding program of Universidade Estadual de Maringá, PR, Brazil, were selected for weight gain. A total of 21 alleles was found in all five polymorphic microsatellite loci (G12292, UNH140; G12311, UNH159; G12312, UNH160; G12314, UNH162; and G12315, UNH163), with an average number between three and five alleles per locus. Allele frequencies ranged from 0.017 (UNH160 - G2) to 0.750 (UNH160 - G0). The average observed heterozygosity was 0.501, 0.391, 0.531, and 0.503 for G0, G1, G2, and G3, respectively. The average inbreeding coefficient was 0.192 (G0), 0.401 (G1), 0.230 (G2), and 0.301 (G3). All generations showed deviation from Hardy-Weinberg equilibrium, with linkage disequilibrium in most loci. Except for G1, the heterozygosity was maintained in the G2 and G3 generations, which indicates that there is no significant loss of genetic variability in the breeding program.
Index terms:Oreochromis niloticus, inbreeding coefficient, allele frequency, tilapia farming, genetic variability.
Introdução
A produção mundial de pescado em 2011 somou 154 milhões de toneladas, dos quais 63,6 milhões foram provenientes da aquicultura (Organización de las Naciones Unidas para la Alimentación y la Agricultura, 2012). No Brasil, esse setor promissor apresentou produção aquícola de 479.398,6 toneladas em 2010, que foi 15,3% superior ao ano 2009 (Boletim estatístico da pesca e aquicultura, 2012).
A tilápia é o segundo grupo de peixes mais produzido no mundo, abaixo somente das carpas (Organización de las Naciones Unidas para la Alimentación y la Agricultura, 2012). Esta condição deve-se à boa qualidade da carne, à facilidade de crescimento e reprodução em diversas condições de cultivo, e à tolerância ao estresse induzido pela manipulação (Tsadik & Bart, 2007). Estas características fazem da tilápia-do-nilo (Oreochromis niloticus) a espécie mais cultivada no Brasil, com produção em 2010 de 155.450,8 toneladas (Boletim estatístico da pesca e aquicultura, 2012).
A intensificação do cultivo tem levado os produtores a buscar linhagens de peixes que apresentem um desempenho superior. Reconhecidos programas de melhoramento genético, como o do salmão do Atlântico (Salmo salar) na Noruega, iniciaram-se com a avaliação de peixes provenientes de 40 rios. A carpa rohu (Cyprinus carpio), na Índia, começou com o cruzamento de seis linhagens diferentes (Toro & López-Fanjul, 2012). Esses exemplos mostram a importância que tem a população-base para obter alta variabilidade genética nesses programas.
A linhagem Gift destaca-se por sua taxa de crescimento rápido, alto rendimento de filé e resistência a doenças. Originária das Filipinas, esta linhagem foi desenvolvida pela organização não governamental WorldFish Center (anteriormente ICLARM), como resultado da seleção e cruzamento de oito linhagens de tilápia: quatro silvestres - do Egito, Gana, Quênia, Senegal; e quatro cultivadas - das Filipinas, Israel, Singapura e Tailândia. O cruzamento e a seleção dessas linhagens foram realizados com o objetivo de aumentar a variabilidade genética e a seleção de características desejadas (Oliveira et al., 2011). A variabilidade genética confere maior capacidade de adaptação a ambientes heterogêneos e permite que as progênies apresentem maior capacidade de se confrontar com variações ambientais (Petersen et al., 2012).
Para selecionar as famílias em um programa de melhoramento é necessário o monitoramento e controle da variabilidade genética ao longo das gerações, a qual pode diminuir em consequência do cruzamento entre indivíduos aparentados (Rutten et al., 2004; Romana-Eguia et al., 2005). A baixa variabilidade genética pode levar ao declínio do desempenho zootécnico, fazendo que características fenotípicas de baixo interesse econômico ou indesejadas sejam expressas (Oliveira et al., 2011). Entre as causas que podem levar à perda de variabilidade genética, se encontram: o inadequado planejamento da reprodução, a diminuição do número efetivo de reprodutores, o cruzamento entre indivíduos muito próximos e as intensas seleções aplicadas (Petersen et al., 2012). Portanto, são necessárias análises genéticas que permitam calcular essa variabilidade no programa de melhoramento, por meio do uso de parâmetros como a heterogozidade, coeficientes de endogamia, frequências alélicas, equilíbrio de Hardy-Weinberg, entre outros. Com esse objetivo, marcadores moleculares microssatélites vêm sendo utilizados com sucesso em pesquisas com populações naturais de peixes e na piscicultura (Romana-Eguia et al., 2004; Rutten et al., 2004; Horreo et al., 2008; Briñez et al., 2011) .
O objetivo deste trabalho foi caracterizar a variabilidade genética dos parentais (G0) e em três gerações consecutivas (G1, G2 e G3) de tilápia-do-nilo, linhagem Gift, por meio de marcadores microssatélites.
Material e Métodos
Em 2005, a Universidade Estadual de Maringá (UEM) recebeu exemplares de 30 famílias da variedade Gift, doadas pela WorldFish Center, o que posicionou o Brasil como o primeiro país da América Latina a implementar o programa de melhoramento genético desta linhagem (Lupchinski, et al., 2008). Características biométricas, desempenho, taxas de mortalidade e idade comercial foram determinadas para o monitoramento e seleção das famílias formadoras das próximas gerações. Essas famílias foram cultivadas em viveiros e denominadas inicialmente de geração zero (G0).
No período de 2007/2008, foi formada a G1 proveniente do cruzamento de 32 famílias de tilápia da G0. Em seguida, realizaram-se cruzamentos de irmãos completos e meios irmãos, para a formação de famílias. No segundo período 2008/2009, foi formada a G2, composta por 58 famílias provenientes da G1 e, em 2009/2010, a G3 foi produto do cruzamento de 78 famílias selecionadas da geração anterior.
Para este trabalho, 360 amostras (90 amostras da G0, G1, G2 e G3) de nadadeira caudal (0,5 cm2 aproximadamente) foram coletadas, armazenadas (álcool etílico 70%) e transportadas até o Laboratório de Biologia Molecular, do Departamento de Zootecnia, da Universidade Estadual de Maringá, onde foram realizadas as análises genéticas.
Para a extração de DNA, utilizou-se a metodologia descrita por Lopera-Barrero et al. (2008). A concentração do DNA foi avaliada em espectrofotômetro, e as amostras foram diluídas para a concentração de 10 ng µL-1. A integridade do DNA foi verificada em gel de agarose a 1% e, posteriormente, a imagem foi capturada pelo sistema de fotodocumentação. Sete iniciadores foram escolhidos do banco de dados National Center for Biotechnology Information (2013): G12261 = UNH168, G12280 = UNH127, G12292 = UNH140, G12311 = UNH159, G12312 = UNH160, G12314 = UNH162 e G12315 = UNH163. Foi observada baixa especificidade e padrão monomórfico nos iniciadores UNH168 e UHN127, os quais foram excluídos das análises. Os cinco loci utilizados são caracterizados na Tabela 1.
O DNA foi amplificado em volume de reação de 16 µL, com 1X tampão Tris-KCl, 2,5 mmol L-1 de MgCl2, 0,8 µmol L-1 de iniciador, 0,4 mmol L-1 de dNTP, 1U de Platinum Taq DNA Polimerase "hot start" e 30 ng de DNA-alvo. A reação em cadeia da polimerase (PCR) foi realizada nas seguintes condições: 4 min de desnaturação a 94ºC; 35 ciclos de 30 s a 94ºC; 30 s a temperaturas específicas de anelamento para cada iniciador (Tabela 1); 1 min a 72ºC; e uma extensão final a 72ºC por 10 min.
Os alelos foram visualizados em gel de poliacrilamida a 10% (acrilamida : bisacrilamida - 29:1), ureia 6 mol L-1, e foram corados com nitrato de prata, conforme Bassam et al. (1991). O tamanho dos alelos foi calculado pelo programa Kodak Edas-290, por meio de escada de DNA (Invitrogen) de 10, 50 e 100 pb.
O número de alelos (Na), a heterozigose observada (Ho) e a esperada (He), o teste do equilíbrio de Hardy-Weinberg (HWE) e o coeficiente de endogamia (Fis) de Wright (1978) foram calculados para cada locus, pelo programa Genepop 4.0.6 (Rousset, 2008). Para os testes de significância das frequências alélicas, He, Ho e Fis, ao longo das gerações, utilizou-se o programa SAS versão 9,0. Para Ho, He e o teste do equilíbrio Hardy-Weinberg (EHW), a deficiência ou excesso de heterozigotos para os loci foi baseado no teste exato de Fisher (a 1% de probabilidade), calculado pelo método de cadeia de Markov (Markov chain length: 100,000; dememorizations: 10,000).
A frequência alélica, os alelos efetivos (Ae) e a distância genética (GD) foram calculados com o programa PopGene 1.31 (Yeh et al., 1999). A presença de alelos nulos foi verificada por meio do programa Micro-Checker 2.2.3 (Van Oosterhout et al., 2004). Os valores de diferenciação genética FST (Weir & Cockerham, 1984), o desequilíbrio de ligação e a análise de variância molecular - Amova (teste exato de Fisher a 5% de probabilidade) foram estimados pelo programa Arlequim 3.0 (Excoffier et al., 2005). Para o FST e Amova, as gerações foram analisadas formando-se todas as combinações possíveis (seis no total). Como método de diferenciação dos valores de FST, foi utilizada a definição de Wright (1978), em que os valores de 0,00 a 0,05, 0,051 a 0,15, 0,151 a 0,25 e >0,25 indicaram pequena, moderada, alta e elevada diferenciação genética. A possível redução do tamanho populacional efetivo foi calculada por meio do programa Bottleneck 1.2.02 (Cornuet & Luikart, 1996), pelos modelos de mutação IAM (modelo de alelos infinito) e de mutação por passos SMM (step-wise mutation) (Estoup et al., 1995). Esses modelos foram usados para evitar a superestimação do excesso ou deficiência de heterozigosidade. A significância foi obtida pelo teste de Wilcoxon, que é mais robusto para amostras com pequeno número de loci polimórficos (<20).
Resultados e Discussão
Do total de 21 alelos observados, os iniciadores UNH159 e UNH163 foram os que apresentaram o maior número (cinco). Os loci UNH140 e UNH160 apresentaram quatro alelos e o locus UNH162 três. O número médio de alelos efetivos variou entre 3,24 (G0) e 3,619 (G3) (Tabela 2). Em geral, o número efetivo de alelos observados foi alto. No entanto, Rutten et al. (2004) e Melo et al. (2006) observaram em suas pesquisas um menor número de alelos nos loci analisados.
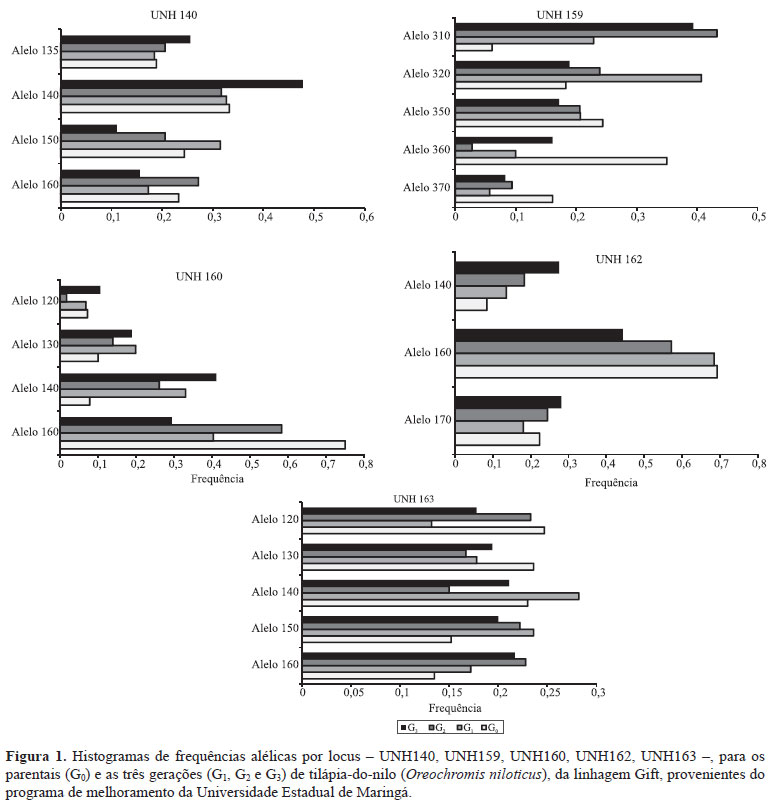
Não foi verificada eliminação de alelo nas gerações. Porém, foram observados vários alelos com baixa frequência (menor que 0,1000 - 10%) nos loci: UNH159/370 pb , em G1, G2 e G3; UNH159/360 pb, em G2; UNH159/310 pb, em G0; UNH160/140 pb, em G0; UNH160/120 pb, em G1, G2 e G3; e UNH162/140 pb, em G0.
Os loci UNH140 e UNH160 não apresentaram diferenças estatísticas nas frequências alélicas ao longo das gerações. No entanto, o locus UNH159 - alelo 350 pb (UNH159/350) apresentou diferença de 10% (p=0,0878) nas gerações, o que evidenciou diminuição da frequência, quando foi gerada a geração seguinte. No locus UNH162/160, também se observou diminuição das frequências, com diferença estatística de 1% (p=0,0135). Contrariamente, UNH159/310 exibiu aumento da frequência (0,33%), desde a G0 à G3, e a diferençaestatísticade 5% (p=0,0474) nas frequências, ao longo das gerações. Também o locus UNH162/140 mostrou aumento da frequência de 0,191%, em comparação à frequência G0 com G3, e a diferença de 5% em todas as gerações (Figura 1).
Uma possível explicação da diferença de frequência nas gerações e da presença de alelos com baixa frequência pode ser a pressão de seleção do programa de melhoramento genético, em que se espera a obtenção de grupos genéticos padronizados. Além disso, o baixo número de alelos pode ser resultante da influência da fixação de alelos, por meio do processo de seleção realizado anteriormente à G0. Romana-Eguia et al. (2005) também observaram alelos de baixa frequência, ao analisar o efeito da seleção em massa no melhoramento da tilápia-do-nilo. Entretanto, estes autores encontraram eliminação alélica na formação das gerações de tilápia, no decorrer da seleção direcional.
Os loci UNH140 e UNH163 apresentaram alelos nulos (Tabela 2), resultantes de mutações ocorridas nas regiões de anelamento dos inciadores. Essa é uma situação muito comum para marcadores microssatélites, que pode levar a erros de genotipagem (Kordicheva et al., 2010).
A média da heterozigosidade observada (Ho), que representa o grau de diversidade genética das gerações, não mostrou diferença nas gerações nem nos loci estudados. Porém a G1 apresentou o menor valor médio 0,391 (Tabela 2). Este valor de Ho pode estar relacionado ao número de famílias usadas para a conformação das gerações (G1 = 32; G2 = 58 e G3 = 78). Este resultado permitiu inferir que a redução do tamanho do plantel reprodutor pode implicar a diminuição da heterozigosidade. Assim, populações que estão submetidas à diminuição radical do tamanho populacional podem evidenciar aumento no coeficiente de endogamia, redução da heterozigose e um processo de deriva genética (Hilsdorf et al., 2006).
Para os valores de heterozigosidade esperada (He), as gerações apresentaram diferença estatística de 3% (p=0,03) no locus UNH162. Este resultado indica que, para a maioria dos loci, tanto para Ho (onde não foram encontradas diferenças) como para He (onde somente um locus apresentou diferença), a variabilidade genética tem se mantido nas gerações. Em contraste, Romana-Eguia et al. (2005), em gerações de tilápia-do-nilo da linhagem Chitralada submetidas à seleção quanto ao ganho em peso, encontraram valores de He significativamente reduzidos da G1 para a G4. Isto mostra que o programa de melhoramento genético da linhagem Gift está mantendo com sucesso a variabilidade genética através das gerações.
O coeficiente de endogamia (Fis) variou de 0,192 (G0) a 0,401 (G1). O locus UNH160 apresentou diferença de 6% (p= 0,06) nas gerações. Na comparação do Fis, a G1 apresentou os maiores valores em três dos cinco loci analisados e o maior valor médio (0,401) (Tabela 2). Os valores positivos do Fis observados nos resultados indicaram a existência de um deficit de heterozigotos a partir da G0. Esse deficit pode estar associado ao estado genético em que se encontravam as famílias vindas da Malásia e ao tipo de seleção e cruzamento utilizados. Assim, a hipótese de que a quantidade de famílias formadoras das gerações interfere nos parâmetros de variabilidade é também sustentada.
No caso das gerações melhoradas de tilápia Gift no Brasil, são realizados acasalamentos hierárquicos (duas fêmeas x um macho) que consideram o pedigree, as características de herdabilidade e os cruzamentos, com base nos valores genéticos das famílias. Mesmo assim, a endogamia é esperada em estoques mantidos em cativeiro (Lopera-Barrero et al., 2010) e pode ser ocasionada pelo uso de um número pequeno de casais como reprodutores (Almeida et al., 2013). Em programas de melhoramento genético, Fis positivos são esperados, já que a intensidade de seleção pode ocasionar deriva genética, ao se reduzir o tamanho efetivo da população originada, dada pela recombinação das progênies selecionadas (Reis et al., 2011). Os valores positivos de Fis ainda podem ser influenciados pela presença de excesso de heterozigotos (Kincaid, 1983); entretanto, os valores de He e Ho do presente estudo não evidenciaram essa condição.
Foram observados desvios significativos em G0 (UNH162), G1 (UNH160 e UNH162) e G2 (UNH159 e UNH162) (Tabela 2). O desequilíbrio de Hardy-Weinberg pode ser causado por diversos motivos, como: endogamia, mistura de várias populações com diferentes frequências de alelos, endocruzamento, excesso de homozigose e presença de alelos nulos (Romana-Eguia et al., 2005; Kordicheva et al., 2010). No presente estudo, o desequilíbrio observado nos resultados pode ser explicado pelo endocruzamento na formação das gerações.
O desequilíbrio de ligação, que representa uma associação entre alelos em dois ou mais loci de uma população (Ramakrishnan, 2013), foi observado em: G0 (UNH159 x UNH162 e UNH160 x UNH163); G1 (UNH140 x UNH159, UNH140 x UNH160 e UNH160 x UNH163); G2 (UNH160 x UNH163); e G3 (UNH140 x UNH160, UNH159 x UNH160 e UNH162 x UNH163). De acordo com Ridley (2006), o desequilíbrio de ligação pode existir em consequência da seleção que favorece indivíduos com combinações particulares de alelos, loci fortemente ligados, deriva aleatória e cruzamento não aleatório, o que leva ao aumento do índice de homozigotos e consequentes desvios de ligação. Porém, mesmo que se tenha encontrado desequilíbrio de ligação para a maioria das combinações dos loci - o que pode estar relacionado à seleção do programa de melhoramento genético -, algumas características como Ho, He e Fis indicaram que as medidas utilizadas como conhecimento do pedigree e cruzamentos, com base em valores genéticos, têm mantido a variabilidade genética do plantel.
A análise de variância molecular e FST, na G0, G1, G2 e G3 (Tabela 3), evidenciaram maior variação dentro das gerações do que entre gerações, com pequena (G0 x G1, G0 xG2), moderada (G0 x G3, G1 x G2) e elevada (G1 x G3, G2 x G3) diferenciação genética. Esses resultados foram corroborados pelos valores de distância genética (Tabela 3) e pelo dendrograma das gerações (Figura 2). Assim, mesmo com a manutenção da variabilidade genética através das gerações, estas podem estar sendo influenciadas pela pressão da seleção, pelo número de famílias e indivíduos selecionados por geração e pelo endocruzamento. Esses fatores podem ocasionar a diminuição de heterozigosidade e a fixação ou perda de alelos no decorrer da seleção.
Em relação à análise bottleneck (efeito gargalo de garrafa), os dois modelos evolutivos analisados (IAM e SMM) identificaram desequilíbrio entre a heterozigosidade esperada (He) e a He em equilíbrio (Heq), para G0 e G1, G2 e G3, o que aponta a ocorrência de um gargalo recente (Tabela 4). Na piscicultura gargalos genéticos podem ocorrer quando apenas alguns casais de reprodutores produzem centenas de descendentes, os quais constituirão toda a geração seguinte (Machado-Schiaffino et al., 2007). Esse procedimento pode também acontecer nos programas de melhoramento genético.
Linhagens melhoradas são essenciais para o crescimento da aquicultura (Lind et al., 2012), e os resultados obtidos no presente estudo apoiam o fato de que métodos de seleção adequados e monitoramento genético devem ser praticados em programas de melhoramento (Romana-Eguia et al., 2005; Moreira et al., 2007; Zhang et al., 2010).
Observou-se que, com exceção da G1, houve manutenção da variabilidade genética nos parentais (G0) e nas gerações (G2 e G3). Isto é importante, ao se considerar que a diminuição da variabilidade genética em animais submetidos ao melhoramento, leva ao decréscimo de algumas características zootécnicas de interesse. Em programas de melhoramento, deve-se procurar manter a variabilidade genética dentro dos plantéis, evitar o acasalamento de indivíduos muito aparentados e sempre utilizar um maior número de famílias para a seleção das próximas gerações.
Conclusão
As frequências alélicas, heterozigosidades e coeficientes de endogamia são mantidos nas gerações G2 e G3 de tilápia-do-nilo, da linhagem Gift, fato que mostra que não há perda significativa de variabilidade genética no programa de melhoramento.
Recebido em 20 de agosto de 2013 e aprovado em 30 de setembro de 2013
- ALMEIDA, F.S. de; LOPES, C.M.; ORSI, M.L.; SIROL, R.N.; SODRÉ, L.M.K. Genetic monitoring by RAPD markers for repopulation programs of Salminus brasiliensis (Pisces, Characiformes). Acta Scientiarum. Animal Sciences, v.35, p.119-126, 2013.
- BASSAM, B.J.; CAETANO, A.G.; GRESSHOFF, P.M. Fast and sensitive silver staining of DNA in polyacrylamide gels. Analytical Biochemistry, v.196, p.80-83, 1991. DOI: 10.1016/0003-2697(91)90120-I.
- BOLETIM estatístico da pesca e aquicultura: Brasil 2010. Brasília: Ministério da Pesca e Aquicultura, 2012. 128p.
- BRIÑEZ, R.B.; CARABALLO, O.X.; SALAZAR, V.M. Genetic diversity of six populations of red hybrid tilapia, using microsatellites genetic markers. Revista MVZ Córdoba, v.16, p.2491-2498, 2011.
- CORNUET, J.M.; LUIKART, G. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data. Genetics, v.144, p.2001-2014, 1996.
- ESTOUP, A.; GARNERY, L.; SOLIGNAC, M.; CORNUET, J.M. Microsatellite variation in honey bee (Apis mellifera L.) populations: hierarchical genetic structure and test of the infinite allele and stepwise mutation models. Genetics Society of America, v.140, p.679-695, 1995.
- EXCOFFIER, L.; LAVAL, G.; SCHNEIDER, S. Arlequin (version 3.0): an integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, v.50, p.47-50, 2005.
- HILSDORF, A.W.S.; RESENDE, E.K. de; MARQUES, D.K.S. Genética e conservação de estoques pesqueiros de águas continentais no Brasil: situação atual e perspectivas. Corumbá: Embrapa Pantanal, 2006. 44p. (Embrapa Pantanal. Documentos, 82).
- HORREO, J.L.; MACHADO-SCHIAFFINO, G.; GRIFFITHS, A.; BRIGHT, D.; STEVENS, J.; GARCIA-VAZQUEZ, E. Identification of differential broodstock contribution affecting genetic variability in hatchery stocks of Atlantic salmon (Salmo salar). Aquaculture, v.280, p.89-93, 2008. DOI: 10.1016/j.aquaculture.2008.05.004.
- KINCAID, H.L. Inbreeding in fish populations used for aquaculture. Aquaculture, v.33, p.215-227, 1983. DOI: 10.1016/0044-8486(83)90402-7.
- KORDICHEVA, S.Y.; RUBTSOVA, G.A.; SHITOVA, M.A.; SHAIKHAEV, G.O.; AFANASIEV, K.I.; ZHIVOTOVSKY, L.A. A search for null alleles at the microsatellite locus of chum salmon (Oncorhynchus keta Walbaum). Russian Journal of Genetics, v.46, p.1019-1022, 2010. DOI: 10.1134/S1022795410080168.
- LIND, C.E.; PONZONI, R.W.; NGUYEN, N.H.; KHAW, H.L. Selective breeding in fish and conservation of genetic resources for aquaculture. Reproduction in Domestic Animals, v.47, p.255-263, 2012. DOI: 10.1111/j.1439-0531.2012.02084.x.
- LOPERA-BARRERO, N.M.; POVH, J.A.; RIBEIRO, R.P.; GOMES, P.C.; JACOMETO, C.B.; LOPES T. da S. Comparación de protocolos de extracción de ADN con muestras de aleta y larva de peces: extracción modificada con cloruro de sodio. Ciencia e Investigación Agraria, v.35, p.77-86, 2008. DOI: 10.4067/S0718-16202008000100008.
- LOPERA-BARRERO, N.M.; RIBEIRO, R.P.; VARGAS, L.; POVH, J.A.; LOPES, T.S.; OLIVEIRA, S.N.; GOMES, P.C. Diversidad genética de Piaractus mesopotamicus utilizado en programas de repoblación. Archivos de Zootecnia, v.59, p.51-62, 2010. DOI: 10.4321/S0004-05922010000100006.
- LUPCHINSKI, E.J.; VARGAS, L.; POVH, J.A.; RIBEIRO, R.P.; MANGOLIN, C.A.; LOPERA-BARRERO, N.M. Avaliação da variabilidade das gerações G0 e F1 da linhagem GIFT de tilápia do Nilo (Oreochromis niloticus) por RAPD. Acta Scientarum. Animal Sciences, v.30, p.233-240, 2008. DOI: 10.4025/actascianimsci.v30i2.4708.
- MACHADO-SCHIAFFINO, G.; DOPICO, E.; GARCIA-VAZQUEZ, E. Genetic variation losses in Atlantic salmon stocks created for supportive breeding. Aquaculture, v.264, p.59-65, 2007. DOI: 10.1016/j.aquaculture.2006.12.026.
- MELO, D.C.; OLIVEIRA, D.A.A.; RIBEIRO, L.P.; TEIXEIRA, C.S.; SOUSA, A.B.; COELHO, E.G.A.; CREPALDI, D.V.; TEIXEIRA, E.A. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v.58, p.87-93, 2006. DOI: 10.1590/S0102-09352006000100013.
- MOREIRA, A.A.; HILSDORF, A.W.S.; SILVA, J.V. da; SOUZA, V.R. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesquisa Agropecuária Brasileira, v.42, p.521-526, 2007. DOI: 10.1590/S0100-204X2007000400010.
- NATIONAL CENTER FOR BIOTECHNOLOGY INFORMATION. National Center for Biotechnology Information [home page] Available at: <http://www.ncbi.nlm.nih.gov/>. Accessed: 11 Dez. 2013.
- OLIVEIRA, S.N.; RIBEIRO, R.P.; LOPERA, N.M.; CANDIOTO, F.B.; RESENDE, E.K. de; LEGAT, A.P. Análise genética de três gerações de tilápia do Nilo (linhagem GIFT) utilizando o marcador RAPD. Acta Scientiarum. Animal Sciences, v.33, p.207-212, 2011. DOI: 10.4025/actascianimsci.v33i2.9202.
- ORGANIZACIÓN DE LAS NACIONES UNIDAS PARA LA ALIMENTACIÓN Y LA AGRICULTURA. Departamento de Pesca y Acuicultura de la FAO. El estado mundial de la pesca y la acuicultura Roma: FAO, 2012. 231p.
- PETERSEN, R.L.; MELLO, G.; GARCIA, J.E.; LIEDKE, A.M.R.; SINCERO, T.C.M.; GRISARD, E.C. Análise da diversidade genética de tilápias cultivadas no Estado de Santa Catarina (Brasil) utilizando marcadores microssatélites. Boletim do Instituto de Pesca, v.38, p.313-321, 2012.
- RAMAKRISHNAN, A.P. Linkage disequilibrium. In: MALOY, S.; HUGHES, K. (Ed.). Brenner's Encyclopedia of Genetics 2nd ed. San Diego: Elsevier, 2013. p.252-253. DOI: 10.1016/B978-0-12-374984-0.00870-6.
- REIS, R.V. dos; OLIVEIRA, E.J. de; VIANA, A.P.; PEREIRA, T.N.S.; PEREIRA, M.G.; SILVA, M.G. de M. Diversidade genética em seleção recorrente de maracujazeiro-amarelo detectada por marcadores microssatélites. Pesquisa Agropecuária Brasileira, v.46, p.51-57, 2011. DOI: 10.1590/S0100-204X2011000100007.
- RIDLEY, M. Evolução 3.ed. Porto Alegre: ArtMed, 2006. 752p.
- ROMANA-EGUIA, M.R.R.; IKEDA, M.; BASIAO, Z.U.; TANIGUCHI, N. Genetic changes during mass selection for growth in Nile tilapia, Oreochromis niloticus (L.), assessed by microsatellites. Aquaculture Research, v.36, p.69-78, 2005. DOI: 10.1111/j.1365-2109.2004.01185.x.
- ROMANA-EGUIA, M.R.R.; IKEDA, M.; BASIAO, Z.U.; TANIGUCHI, N. Genetic diversity in farmed Asian, Nile, and red hybrid tilapia stocks evaluated from microsatellite and mitochondrial DNA. Aquaculture, v.236, p.131-150, 2004. DOI: 10.1016/j.aquaculture.2004.01.026.
- ROUSSET, F. Genepop'007: a complete re-implementation of the GENEPOP software for Windows and Linux. Molecular Ecology Resources, v.8, p.103-106, 2008. DOI: 10.1111/j.1471-8286.2007.01931.x.
- RUTTEN, M.J.M.; KOMEN, H.; DEERENBERG, R.M.; SIWEK, M.; BOVENHUIS, H. Genetic characterization of four strains of Nile tilapia (Oreochromis niloticus L.) using microsatellite markers. Animal Genetics, v.35, p.93-97, 2004. DOI: 10.1111/j.1365-2052.2004.01090.x.
- TORO, M.A.; LÓPEZ-FANJUL, C. Diseño de programas y mejora genética. In: PORTELA, P.M.; HUERTAS, A.F. (Ed.). Genética y genómica en acuicultura Madrid: Fundación Observatório Español de Acuicultura, 2012. p.185-211. (Publicaciones científicas y tecnológicas del Observatório Español de Acuicultura).
- TSADIK, G.G.; BART, A.N. Effects of feeding, stocking density and water-flow rate on fecundity, spawning frequency and egg quality of Nile tilapia, Oreochromis niloticus (L.). Aquaculture, v.272, p.380-388, 2007. DOI: 10.1016/j.aquaculture.2007.08.040.
- VAN OOSTERHOUT, C.; HUTCHINSON, W.F.; WILLS, D.P.M.; SHIPLEY, P. Micro-Checker: software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, v.4, p.535-538, 2004. DOI: 10.1111/j.1471-8286.2004.00684.x.
- WEIR, B.S.; COCKERHAM, C.C. Estimating F-statistics for the analysis of population structure. Evolution, v.38, p.1358-1370, 1984. DOI: 10.2307/2408641.
- WRIGHT, S. Evolution and the genetics of populations: variability within and among natural populations. Chicago: The University of Chicago, 1978. v.4, 580p.
- YEH, F.C.; BOYLE, T.Y.Z.; XIYAN, J.M. PopGene Version 131: Microsoft Window-based freeware for population genetic analysis. Alberta: University of Alberta and Center for International Forestry Research, 1999. 29p.
- ZHANG, T.; KONG, J.; WANG, W.; WANG, Q. Genetic variability assessed by microsatellites in the breeding populations of the shrimp Penaeus (Fenneropenaeus) chinensis in China. Aquaculture, v.310, p.229-233, 2010. DOI: 10.1016/j.aquaculture.2010.07.025.
Datas de Publicação
-
Publicação nesta coleção
17 Jan 2014 -
Data do Fascículo
Out 2013
Histórico
-
Recebido
20 Ago 2013 -
Aceito
30 Set 2013